本記事では羊土社から発売されている「独習Pythonバイオ情報解析」という教科書から学んだことをアウトプットします。
この本は、Pythonを用いてバイオ情報解析に欠かすことができない、
- データの取扱、整理
- データの可視化
- 統計
- 主成分分析などの次元削減
- クラスタリング
などが解説されています。
私自身、ベースは組織工学で基礎生物学とは少し離れたところにいるので、次元削減やクラスタリングは論文で見かける頻度が増えてきているものの、読み解く解像度が低いことが悩みでした。
この本では自分が普段触れているPythonを使って、バイオ系でどんどん発展している遺伝子解析などついての解析手法を基礎から応用まで解説しているとのことで、自分の悩みを解決してくれそうとのことで手に取りました。
1冊通して学んでみると、基礎・原理・応用をとってもわかりやすく解説されている非常にクオリティの高い教科書でした。
- これからPythonを使ってみたいバイオ系の人
- Pythonは既に使っているけれど、バイオ系の解析はこれから学んでみたい人
- 論文で見る主成分分析、クラスタリングの解釈の解像度を高めたい人
そんな人たちに是非ともおすすめしたい1冊でした。
「独習Pythonバイオ情報解析」を読んだきっかけ
この教科書を手にとったきっかけは、次元削減やクラスタリングなどのデータを使うというよりは理解する解像度を高めたいというものです。
その理由は、最近組織工学の分野でも次元削減やクラスタリングなどを用いたデータが増えてきており、データの解釈の解像度が低いことが悩みでした。
データの解釈はその事象を正確に捉える、疑問を持つという点において非常に重要な能力になるので、データの解釈の解像度を上げる努力は怠りたくありません。
他の理由としては、実際に仕事でもRNA-Seqを用いた解析に触れることが出てきたからというのもあります。
基本的には便利なWebツールを使うことと、ベースがRということで、Pytonを使うことはありませんが、「内部でどんなことをやっているのか」を理解しないとなかなか出てきたデータを信用して解釈するのが難しいのでその理解の助けになるのではというところもこの教科書を手にとった理由になります。
実際に読んで非常に理解の助けになる良い教科書でした。
「独習Pythonバイオ情報解析」で解説されていること
この教科書で解説されている内容としては下記の通りになります。
第1章 この本の使い方と事前準備
「独習Pythonバイオ情報解析」、目次より
第2章 Jupyter Notebokの使い方
第3章 Python速習コース
第4章 文字列処理の基本-ファイルの読み書き、正規表現-
第5章 Biopythonを用いた塩基配列データの扱い方-オブジェクト指向入門-
第6章 pandasはじめの一歩-表敬式データの扱い方-
第7章 RNA-Seqカウントデータの処理-pandas実践編-
第8章 データの可視化-Matplotlib, Seabornを用いたグラフ作成-
第9章 統計的仮説検定-RNA-Seqデータを用いた検定の基本からモデル選択まで-
第10章 シングルセル解析①-テーブルデータの前処理-
第11章 シングルセル解析②-次元削減-
第12章 シングルセル解析③-クラスタリング-
付録A Nunpy入門
付録B Scanpyを使ったシングルセル解析
Pythonを使ったことがない人でも、導入方法からしっかり解説されているため、安心して内容を読み進めることができます。
Pythonを使ったことはまだないけど興味がある人が、実際にPythonを使ってどんな解析ができるのかを学びながらPythonを知るための教科書としても使えます。
この遺伝子などの解析は基本的にはRでやることのほうが圧倒的に多いとのことですが、自分のように、そこまで本格的に解析はしていない、でも論文を読むときにデータの解釈をしっかりできるようになりたいというときに、Pythonを通して原理の部分にふれることができるのがとっても良いと感じました。
自分のようにRよりもPythonの方に馴染みがある人から、バイオの解析について触れることができるということで非常にありがたい1冊です。
そして、一通りの解析の基礎に触れながらも、それぞれの解析では
- 何を見ているのか
- どう解釈するべきか
- 何を注意しなければいけないのか
など原理から応用までしっかりとカバーされていて、体系的にまとめられているため、ネットの解説記事をまばらに読んでいくよりもかなり理解が進みました。
自分が特に参考になった内容
次元削減
基礎生物系や医学系以外の組織工学系などの論文でも目にすることが多くなってきた主成分分析を始めとした下のような図があります。
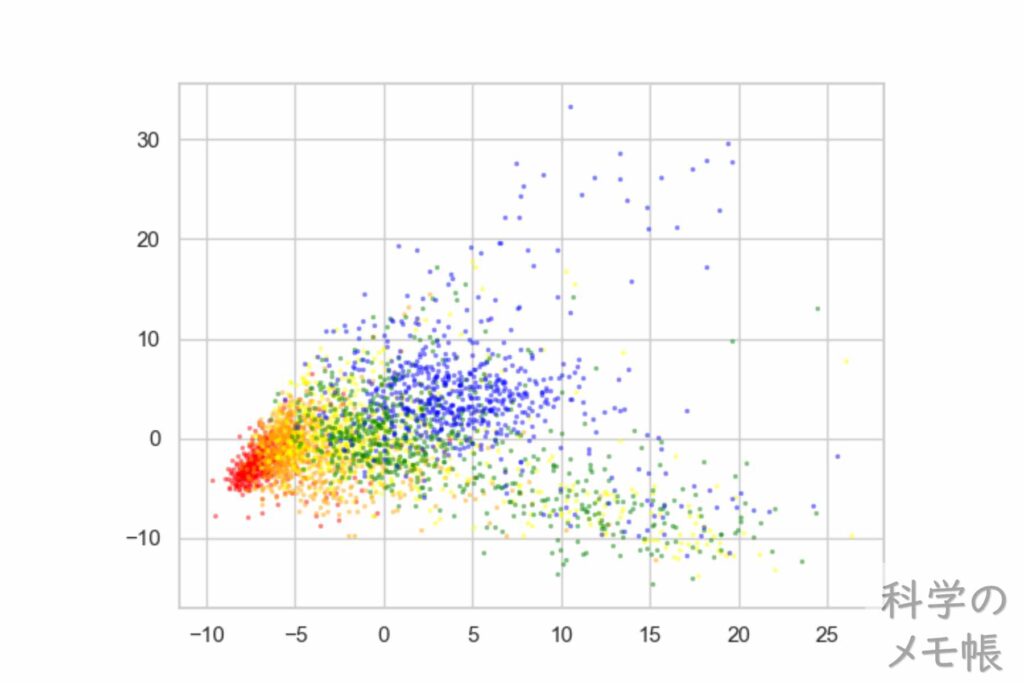
この教科書を通して学習すれば、こんな感じの図が書けるようになり、解釈するための方法がすごく丁寧に解説されていました。
次元削減と一言で言っても、
- 主成分分析
- t-SNE
- UMAP
などの表現方法があり、更に新しい手法も増えているようで、これらは一体何をどうやって表現しているの?というところがなかなかつかめませんでした。
主成分分析は結構使う場面が増えてきており、自分にとっても馴染みのあるものになってきていますが、t-SNEやUMAPは論文で見ていざ調べてみても次元削減して集団の解釈をしやすくしているぐらいの理解しかできていませんでした。
実際に、この教科書で実践して解説を読んで「それぞれのプロットの結果が何を表現しているのか、何を意味しているのか」について自分が納得できるレベルで理解が進んだように思います。
また、主成分分析自体はバイオ以外でも用いられているとのことで、ここで使い方を覚えておくと今後かなり役立つ便利なツールを入手できるチャンスになります。
クラスタリング
これも基礎生物から少しずれた組織工学系の論文でも目にする機会が増えてきているように感じます。
なんとなく表現したいことはわかるのだけれど、どうやって解析しているんだろうと疑問に思っていたものになります。
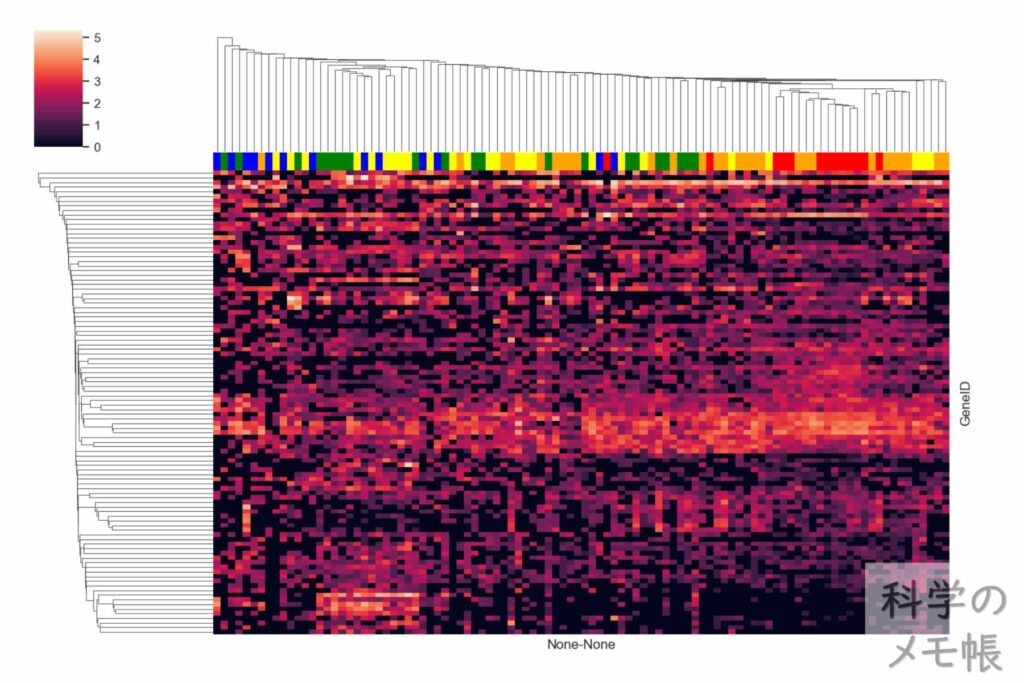
これもpythonを使ってきれいにぱぱっと出力することができます。
(Rのほうが簡単とか言わない)
このクラスタリングも解釈するためには、どんな手法、計算で結果を出力しているのかを知っておいたほうが理解が進むと思うのですが、これも主成分分析同様しっかり解説されていました。
実際に使う機会は少ないにせよ、論文などのデータを読み解く解釈の解像度がかなり上がりました。
特に、最後に「結局どれを使えばいいのか」という解説があり、
“データと目的による”
“どの手法もどのパラメータも正しくはない。どれも間違っている。しかし全てが間違っているわけでもなく、真実の一側面を反映している。”
「独習Pythonバイオ情報解析」, p372-p373
という部分が、このようなデータを読み解くためのツールを使う姿勢、考え方として参考になりました。
まとめ
以上、「独習Pythonバイオ情報解析」から学んだことのアウトプットでした。
Pythonの最低限の基礎からバイオ情報解析手法の難しいところまで体系的にまとめて、しかも原理も押さえられていて、かなりクオリティの高い教科書だと思いました。
- これからPythonを使ってみたいバイオ系の人
- Pythonは既に使っているけれど、バイオ系の解析はこれから学んでみたい人
- 論文で見る主成分分析、クラスタリングの解釈の解像度を高めたい人
そんな人たちに是非ともおすすめしたい1冊です。
Python関係では色々な教科書を使ってきましたが、1冊通して学んだときの充実感が非常に大きかったです。
6000円とそれなりに高価な教科書ではありますが、その価値以上はあると思いました。
情報解析系の情報はネットで入手できますが、プロの人達が試行錯誤して体系的にまとめた教科書の価値を改めて感じる素晴らしい一冊です。





コメント